- 服務
- ·
RNC-seq
- 產品介紹
- 案例解析
- 結果展示
- 送樣建議
服務介紹
對與核糖體結合的正在翻譯的全長RNA進行測序,可以進一步深化轉錄組研究,直接在翻譯水平對應生物表型,可知哪些基因正在翻譯,以及翻譯起始效率;可通過翻譯機制研究彌補轉錄組測序差異表達基因過多或過少的問題;同時也可以分析來源于circRNA或lncRNA的ORF,預測可能翻譯的circRNA,且較之Ribo-seq捕獲的短片段具有更高的靈敏度,能更容易獲得circRNA的信號。
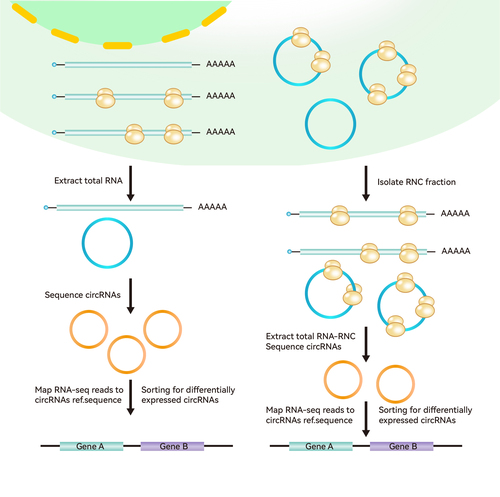
核糖體-新生肽鏈復合物測序技術路線
測序方案
測序平臺:Illumina Novaseq 6000/NovaSeq X Plus
測序模式:PE150
測序數據量:10 Gb raw dataA peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma
NATURE COMMUNICATIONS(2018),DOI: 10.1038/s41467-018-06862-2
測序策略:RNA-seq + RNC-seq
該研究對人的正常細胞和膠質瘤細胞分別進行轉錄組測序(RNA-seq)及翻譯組測序(RNC-seq),通過測序數據分析,總共識別了15189個circRNA分子,其中7017個來自RNA-seq數據,12863個來自RNC-seq數據。對兩組樣本數據中顯著差異表達的circRNA分子取交集,初步篩選出了320個circRNA分子(圖1)。由于大部分circRNA的CDS區域與其編碼蛋白的母基因相同,為了排除假陽性數據的干擾,研究團隊主要關注來源于非編碼基因的10個候選circRNA分子。進一步通過蛋白編碼能力預測發現了5個具有蛋白編碼能力的circRNA分子。最終研究團隊選擇來源于LINC-PINT基因的circRNA進行后續研究。該circRNA可以編碼一個87個氨基酸的多肽(圖2),這個多肽可直接與聚合酶相關因子復合物(PAF1c)相互作用,抑制多癌基因的轉錄延伸。該多肽及其相應的circRNA表達水平在膠質母細胞瘤中較正常組織降低。研究結果證實了circRNA編碼的肽段的存在及其在膠質母細胞瘤發生發展中的潛在功能。
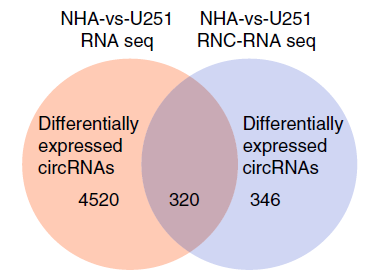
Figure1. 兩種測序顯著差異表達的circRNA分子取交集
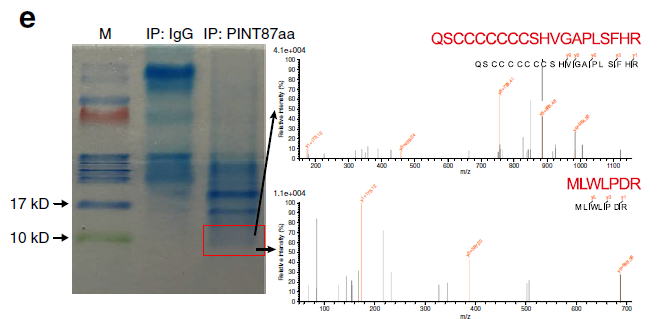
Figure2. 特異性抗體IP實驗及LC-MS/MS分析鑒定PINT87aa多肽序列
生物信息分析
基礎分析
原始數據質控檢查
比對結果質控檢查
基因覆蓋度分析
基于基因表達的樣品主成分分析
基因表達分析
差異基因表達分析
差異基因GO功能分析
差異基因KEGG通路分析
差異基因Rectome通路分析
針對mRNA基因的GSEA分析
circRNA預測及鑒定
circRNA差異分析
差異circRNA宿主基因的GO功能分析
差異circRNA宿主基因的KEGG通路分析
差異circRNA宿主基因的Rectome通路分析
差異circRNA的靶向miRNA預測分析
差異circRNA及靶向miRNA網絡調控制圖
長鏈非編碼RNA的識別
長鏈非編碼RNA差異分析
差異lncRNA順式調控基因的GO功能分析
差異lncRNA順式調控基因的KEGG通路分析
差異lncRNA順式調控基因的Rectome通路分析
高級分析
circRNA翻譯潛能預測分析
翻譯效率分析
*可選擇只測circRNA的方法步驟進行實驗,詳詢技術支持
部分結果示例
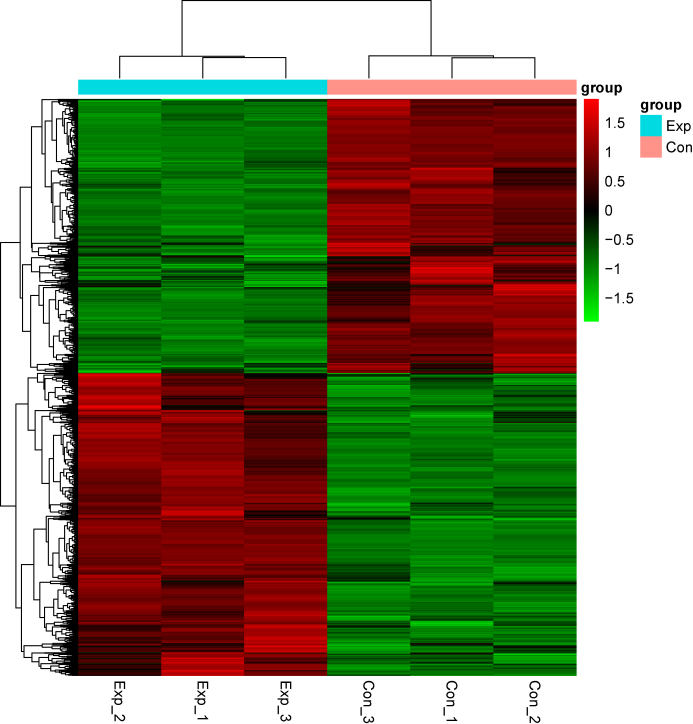
圖1. RNC-seq差異表達基因熱圖展示
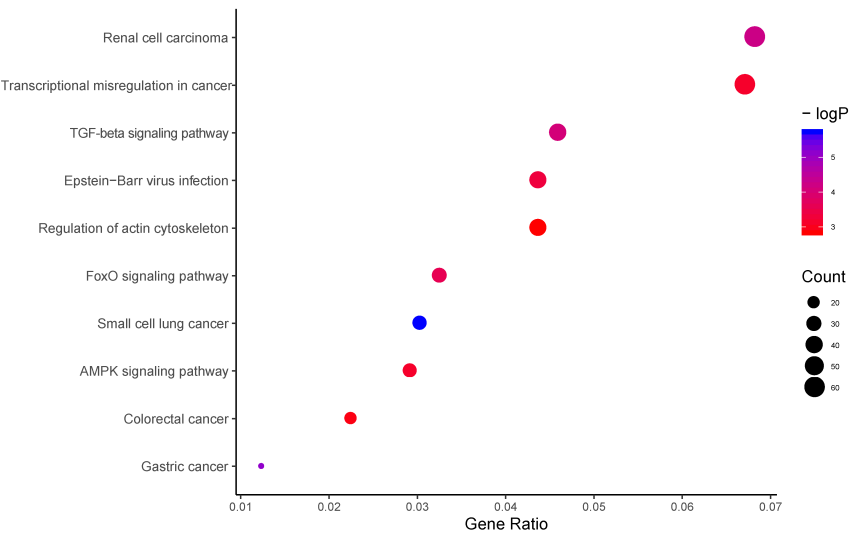
圖2. RNC-seq差異表達circRNA的KEGG通路富集圖展示

圖3. RNC-seq的circRNA-miRNA網絡圖展示
Table1. RNC-seq原始樣本送樣建議
送樣類型
送樣量
備注
細胞(細胞數)
≥1*10^7
無支原體污染
動物組織
≥100mg
植物組織
≥100mg
*翻譯組測序的細胞樣本處理方法和轉錄組測序不一樣,具體處理、保存和運輸方法請咨詢技術支持。
物種范圍:人、小鼠、大鼠,其他物種請詳詢銷售或技術支持
填寫需求描述給我們
工具快速咨詢
400-8989-400
geneseed@geneseed.com.cn

 購物車
購物車




 廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓
廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn