- 服務
- ·
MeRIP-Seq
- 產品介紹
- 案例解析
- 結果展示
- 送樣建議
- FAQ
服務介紹
通過m6A特異性抗體識別并結合具有m6A修飾的RNA片段進行免疫共沉淀,對富集下來的RNA片段進行高通量測序。結合生物信息學分析,即可在全轉錄組范圍內對m6A修飾進行系統研究。
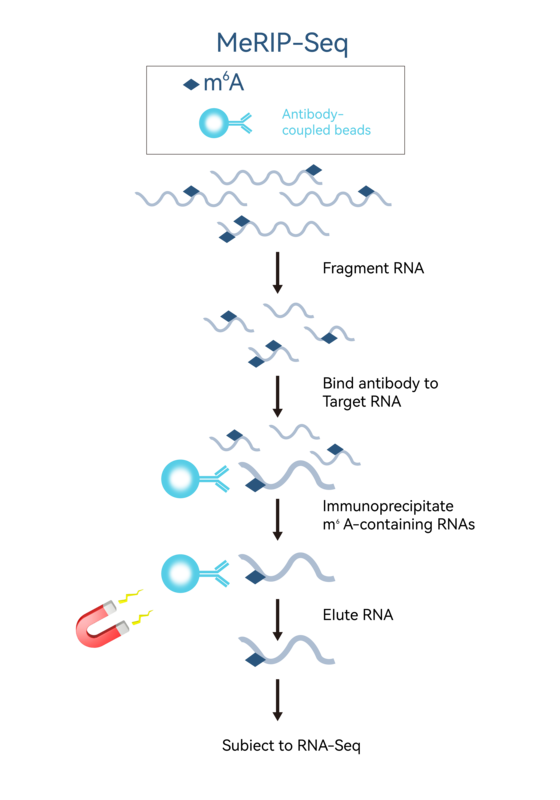
RNA m6A甲基化測序技術路線
測序方案
上機平臺:Illumina Novaseq 6000/NovaSeq X Plus
測序模式:PE150
測序數據量:12 Gb raw data案例1
案例2
生物信息分析
基礎分析
測序數據質量評估
比對結果質控
基因覆蓋度分析
m6A peaks識別
m6A peaks特征注釋
m6A mRNA分析
甲基化mRNA的motif分析
m6A lncRNA分析
甲基化lncRNA的motif分析
circRNA識別鑒定分析
circRNA的m6A富集分析
甲基化circRNA的motif分析
高級分析
甲基化基因的差異表達分析
差異甲基化基因GO功能富集分析
差異甲基化基因KEGG通路分析
差異甲基化基因reactome通路分析
甲基化lncRNA的差異表達分析
差異甲基化lncRNA的順式編碼基因的GO功能富集分析
差異甲基化lncRNA的順式編碼基因的KEGG通路分析
差異甲基化lncRNA的順式編碼基因的Reactome通路分析
甲基化circRNA的差異表達分析
差異甲基化circRNA宿主基因的GO功能富集分析
差異甲基化circRNA宿主基因的KEGG通路分析
差異甲基化circRNA宿主基因的Reactome通路分析
個性化分析
與轉錄組關聯分析
關聯分析四象限圖
部分結果示例

圖1. RIP 富集分布分析
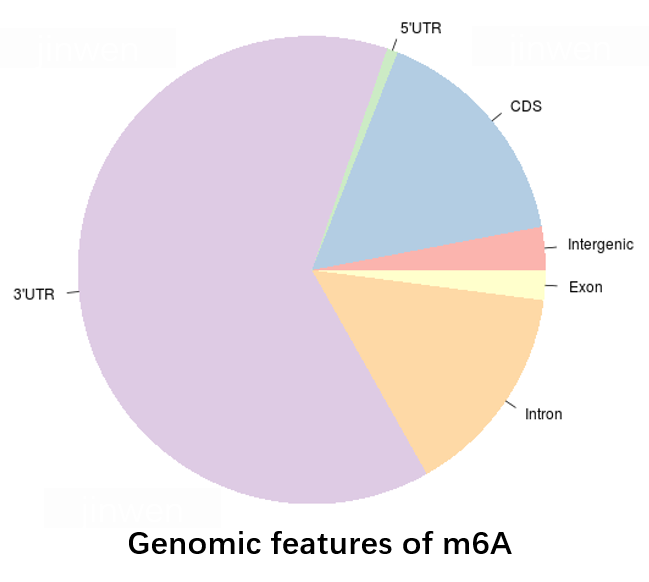
圖2. m6A基因組特征分布分析
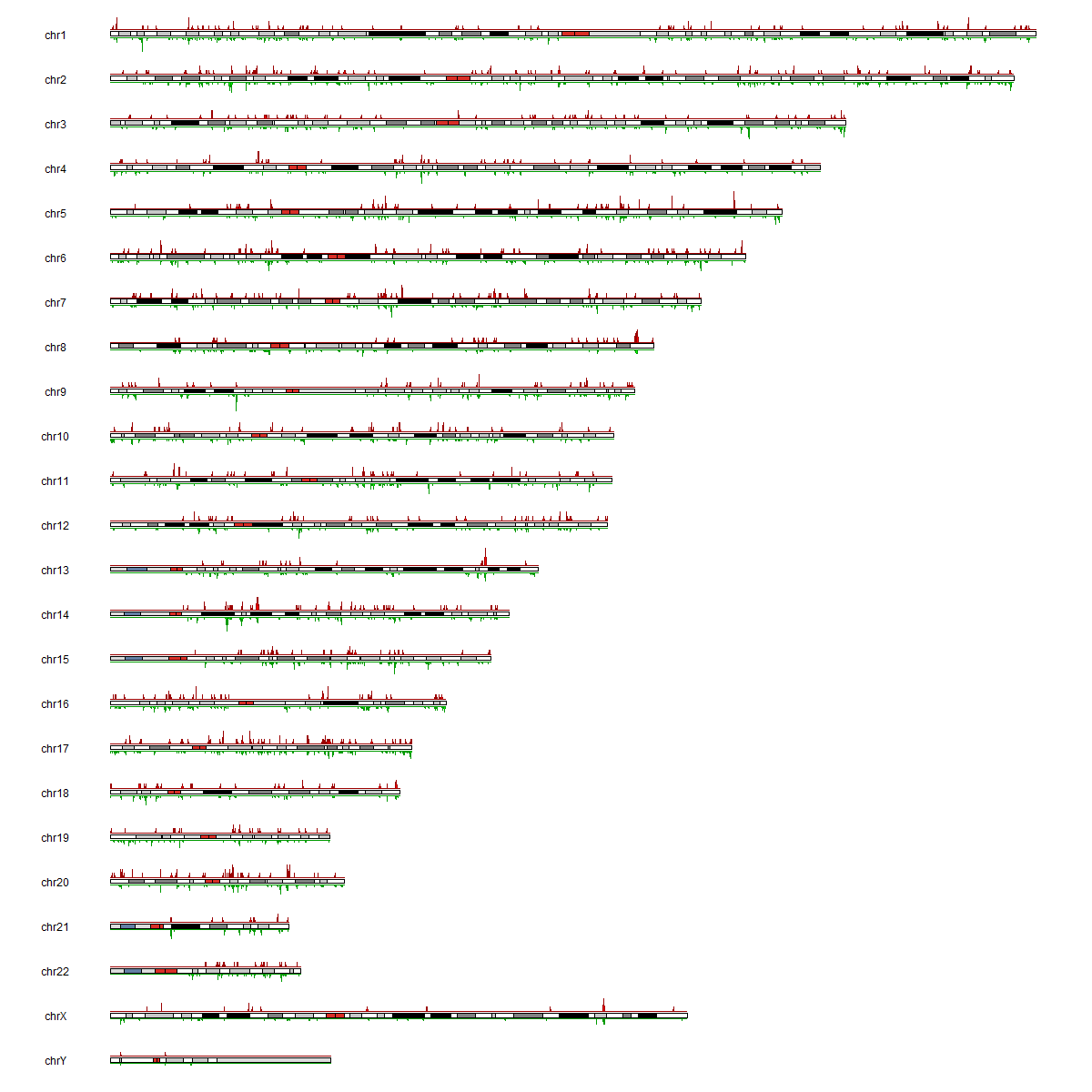
圖3. m6A 全基因組分布分析
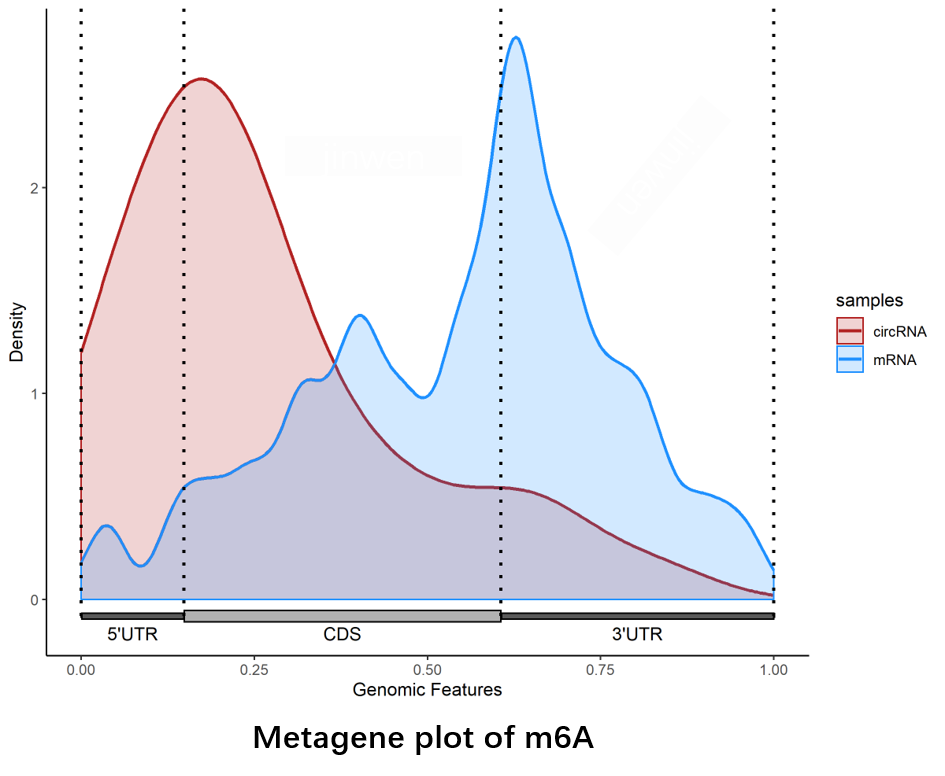
圖4. m6A的元基因分析
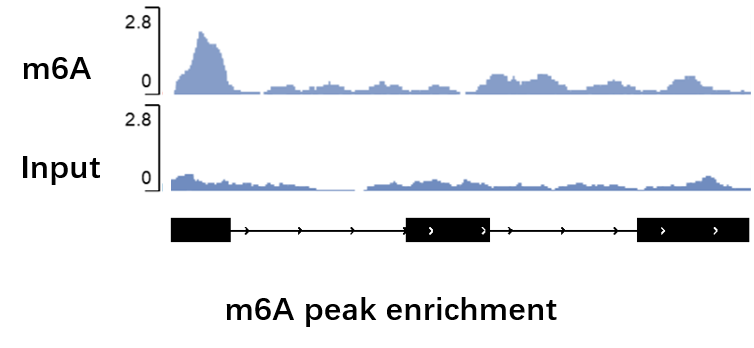
圖5. m6A 特定基因豐度展示
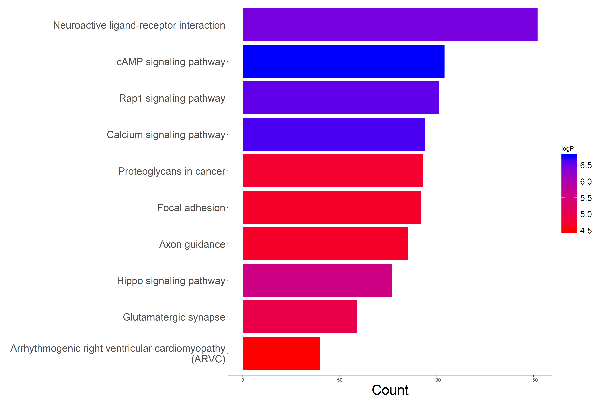
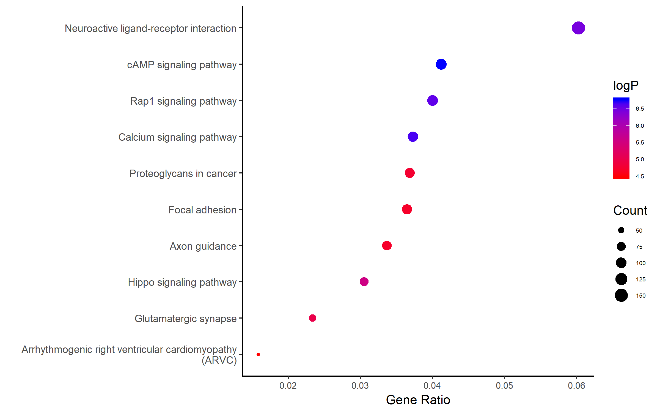
圖6. KEGG通路富集分析
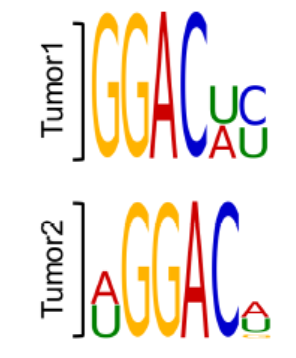
圖7. Motif分析
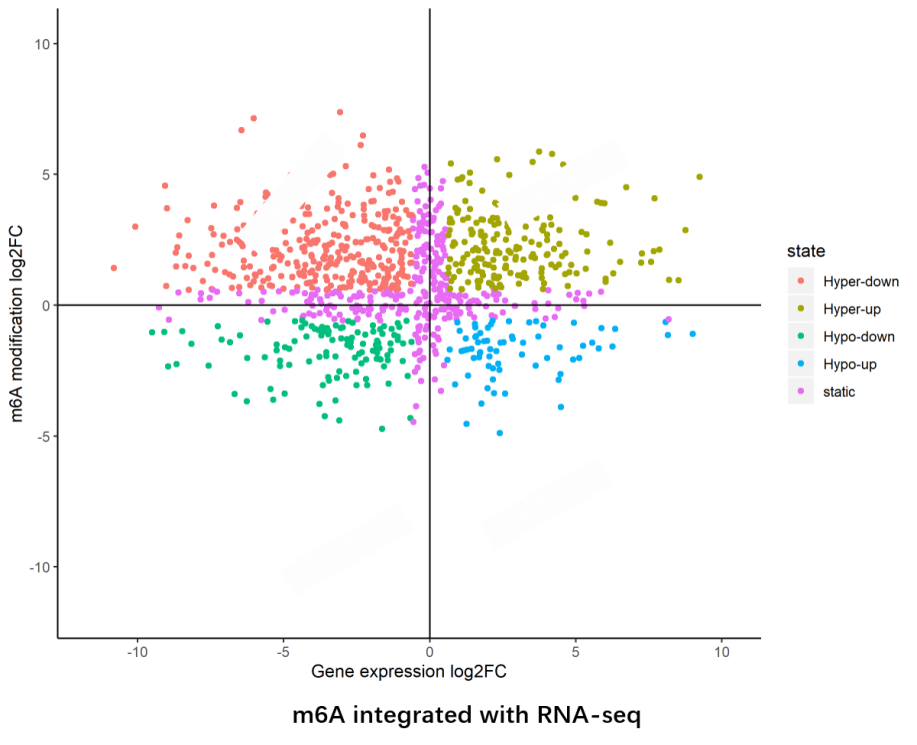
圖8. 轉錄組聯合分析四象限圖
Table.MeRIP-Seq原始樣本送樣建議
送樣類型
送樣量
備注
細胞 (細胞數)
≥5*10^7
無支原體污染
動物組織
≥300mg
植物組織
≥300mg
Table.MeRIP-Seq RNA樣本送樣建議
送樣類型
送樣量
完整性(RIN值)
濃度
純度
total RNA
≥200μg
≥7
≥100ng/μl
無DNA,蛋白/鹽離子等污染,樣本無色透明不粘稠
*更具體的送樣方法請詳詢銷售或技術支持
物種范圍:人、小鼠、大鼠等哺乳動物,其他物種請咨詢銷售或技術支持
Q1:做MeRIP-seq是否還需要另外做普通的轉錄組測序?
A1:不需要,Input數據即可作為普通全轉錄組測序數據進行分析。
Q2:為什么MeRIP-seq需要同時測Input和IP樣本?
A2:MeRIP-seq實驗設置中Input和IP組成一對樣品。實驗環節IP樣品用m6A抗體特異性富集甲基化修飾的RNA片段,而Input僅僅是片段化的RNA則作為對照消減背景噪音,在建庫、測序平行開展。結合峰檢測分析需整合兩個樣本的數據,并利用Input數據排除本底表達水平高或非特異性結合的peaks,以提高calling peak的準確性。
Q3:開展MeRIP-seq是否有物種限制?
A3:如不是人、小鼠和大鼠的物種建議先進行咨詢。一般有參考基因組,且基因組拼接至染色體水平,gtf注釋文件較完整的物種都可以開展;真核、原核或病毒在結合峰檢測涉及的軟件和參數存在差別,原核或病毒項目建議先評估。
填寫需求描述給我們
工具快速咨詢
400-8989-400
geneseed@geneseed.com.cn

 購物車
購物車




 廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓
廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn