- 服務
- ·
mRNA-seq
- 產品介紹
- 結果展示
- 送樣建議
- FAQ
服務介紹
通過oligo-dT磁珠捕獲帶polyA尾的RNA進行建庫測序,可研究mRNA差異表達或檢測結構變異,篩選與疾病或性狀相關的分子標記,還可揭示轉錄組的復雜性,確定基因以及轉錄本結構、可變剪接、RNA編輯、帶polyA尾的非編碼RNA和新轉錄本。
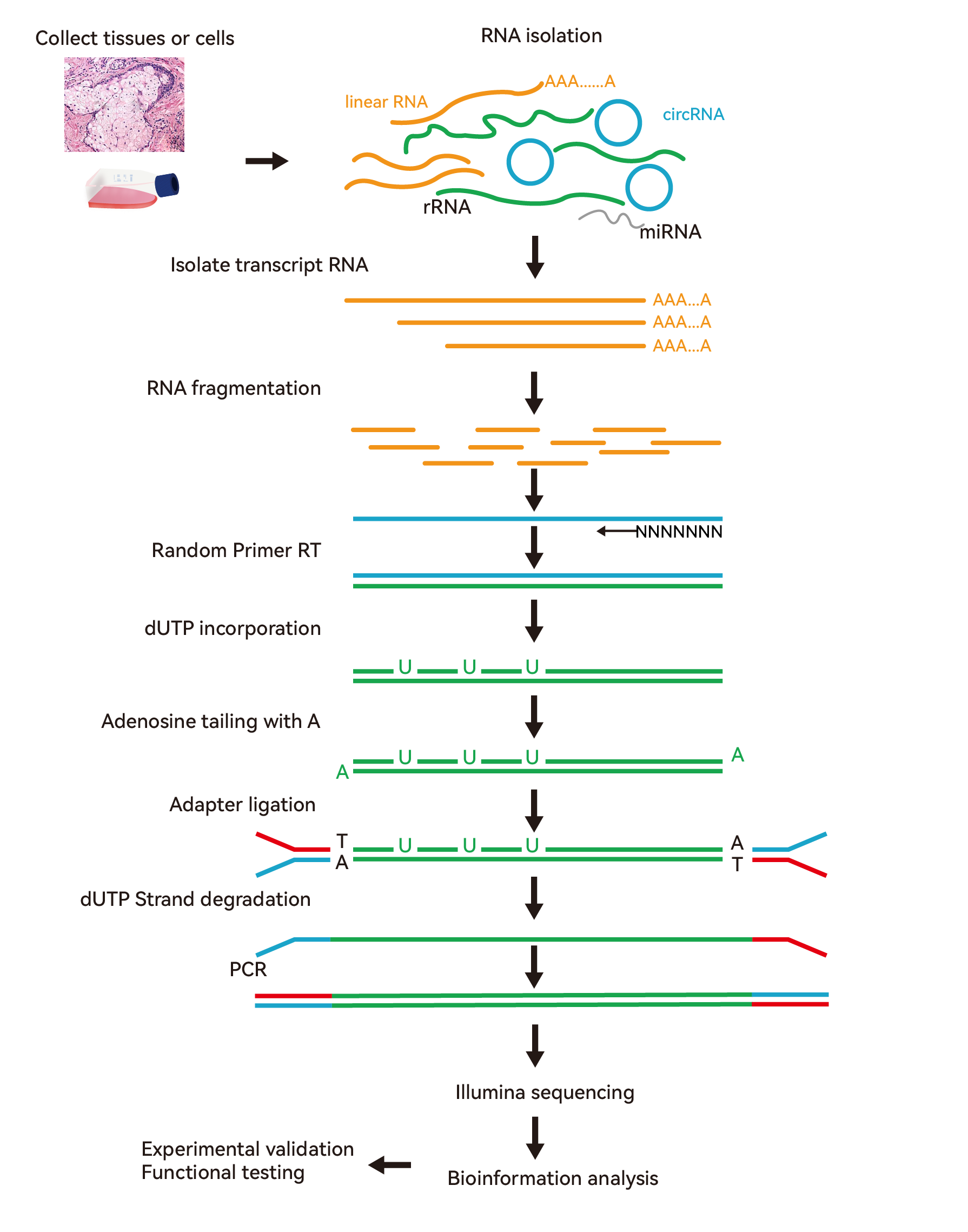
mRNA測序技術路線
測序方案
上機平臺:Illumina Novaseq 6000/NovaSeq X Plus
測序模式:PE150
測序數據量:5 Gb raw data
生物信息分析
原始數據質控檢查
比對結果質控檢查
基因覆蓋度分析
基于基因表達的樣品主成分分析
基因表達分析
差異基因表達分析
差異基因GO功能分析
差異基因KEGG通路分析
差異基因Rectome通路分析
針對mRNA基因的GSEA分析
可選分析
可變剪切分析(測序數據量為12Gb)
部分結果示例
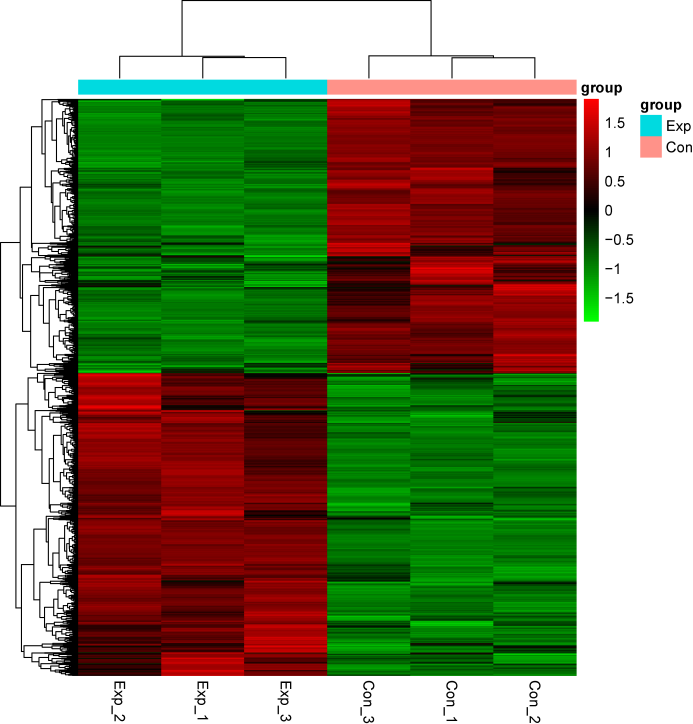
圖1.差異表達基因熱圖展示
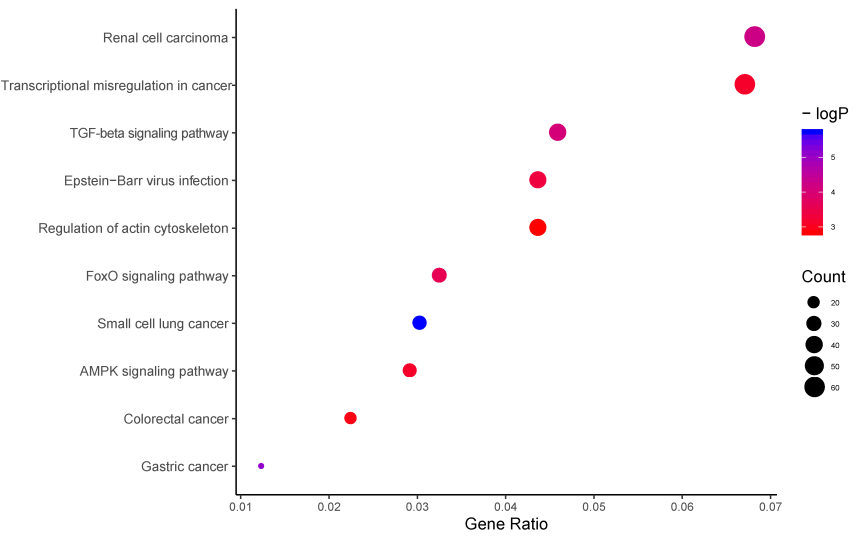
圖2.差異表達基因KEGG通路富集圖展示
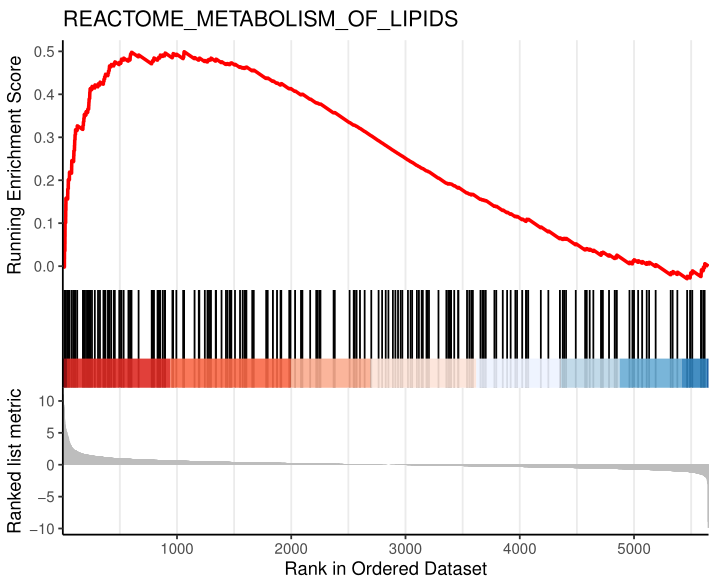
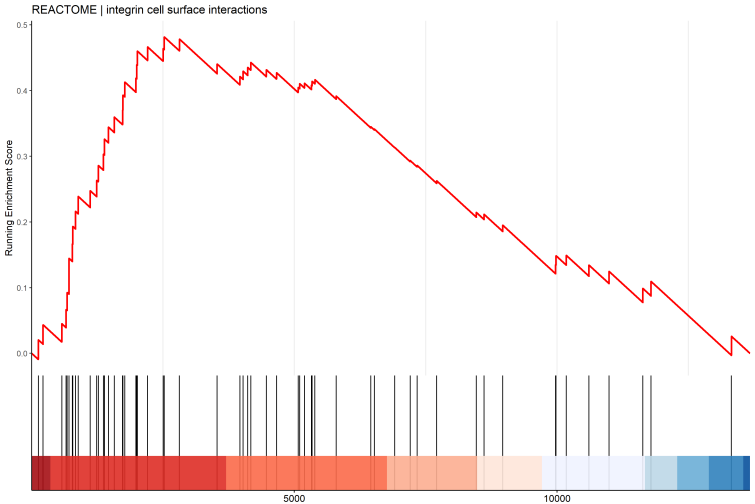
圖3. mRNA的GSEA分析毛毯圖展示
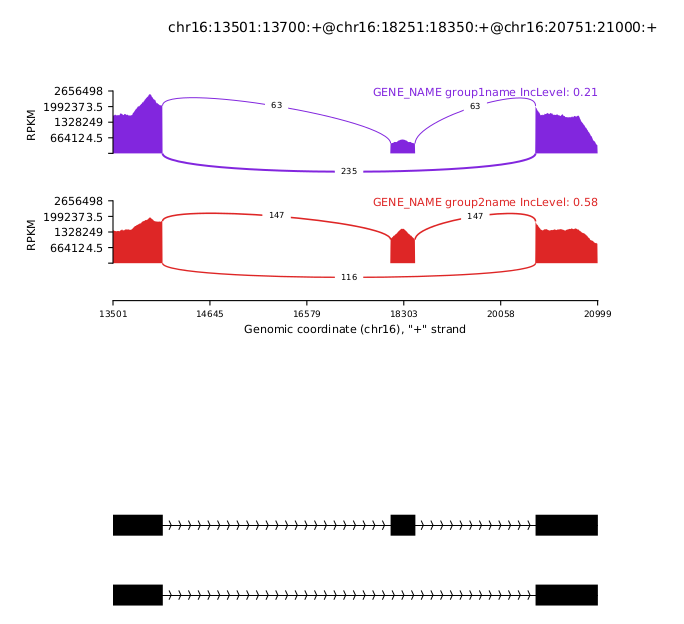
圖4. 單個可變剪切事件sashimi plot
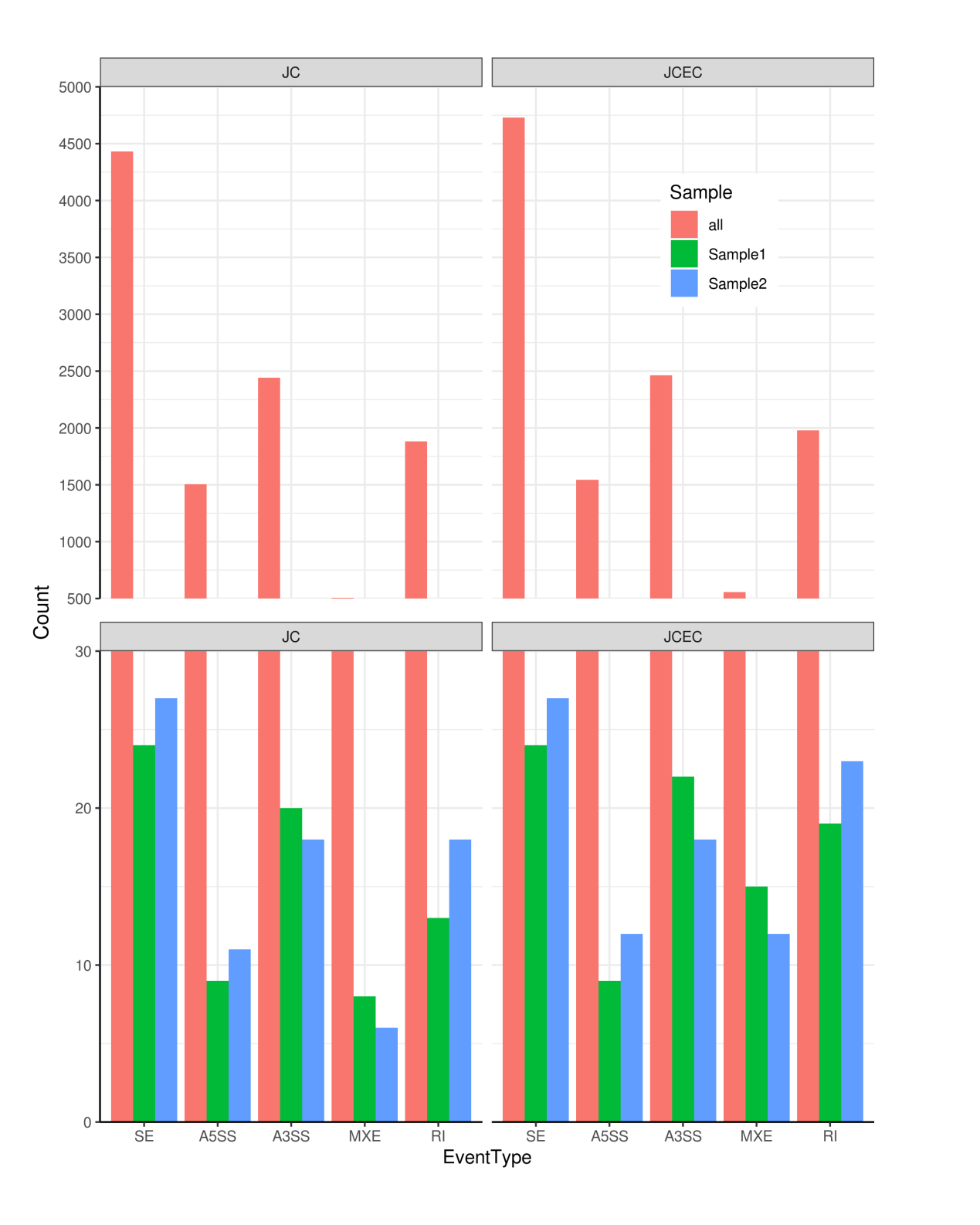
圖5. 可變剪切事件數目統計展示
Table1. mRNA-seq原始樣本送樣建議
送樣類型
送樣量
備注
細胞 (細胞數)
≥1*10^6
無支原體污染
動物組織
≥100mg
植物組織
≥100mg
全血
≥4mL
Table2. mRNA-seq RNA樣本送樣建議
送樣類型
送樣量
完整性(RIN值)
濃度
純度
total RNA(組織、細胞、全血等)
≥2μg
≥8
≥100ng/μl
無DNA,蛋白/鹽離子等污染,樣本無色透明不粘稠
*更具體的送樣方法請咨詢銷售或技術支持
物種范圍:人、小鼠、大鼠,擬南芥、水稻、番茄、玉米、大豆等真核生物,其他物種詳詢銷售或技術支持
Q1:所有真核生物都能做mRNA測序嗎?
A1:真核生物mRNA含有poly-A尾,我們目前常用于富集真核生物mRNA的方法就是采用附著poly-T oligo的磁珠從total RNA 中抓取mRNA,進行建庫測序。所以理論上說,所有的真核生物都能進行mRNA測序。
如果是非常規物種,建議客戶提供物種的拉丁名,進行評估,如物種參考基因組的注釋信息足夠開展分析,是可以做的。
Q2:為什么mRNA測序對RNA的完整性要求比全轉錄組測序的高?
A2:mRNA測序建庫是用磁珠捕獲帶polyA尾的RNA(大部分是mRNA)進行建庫的,如果RNA的完整性較差,部分mRNA降解,polyA尾斷開,能捕獲到的mRNA相對完整性好的樣本要少,基因的覆蓋度相對就較差。因此對RNA的完整性要求更高。
Q3:mRNA-seq 是否可以檢測非編碼 RNA?
A3:mRNA-seq 主要捕獲帶有 poly(A) 尾巴的 RNA 分子,因此,所有帶有 poly(A) 尾巴的分子理論上只要在細胞中表達都有可能被檢測到,主要包括:1)mRNA;2)帶 poly(A) 尾的 lncRNA。
填寫需求描述給我們
工具快速咨詢
400-8989-400
geneseed@geneseed.com.cn

 購物車
購物車




 廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓
廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn