- 產品
- ·
RNase R
- 產品介紹
- 案例分析
- 客戶文章
- 說明書下載
- Q&A
RNase R是一種來源于大腸桿菌RNR超家族的3’-5’核糖核酸外切酶,可從3’-5’方向將RNA逐步切割成二核苷酸和三核苷酸。RNase R可消化幾乎所有的線性RNA分子,但不易消化環形RNA、套索結構或3’突出末端少于7個核苷酸的雙鏈RNA分子。
RNase R是circRNA的鑒定和富集實驗必備工具酶,可消化線性RNA以使環形RNA(circRNAs)或套索結構RNA(lariat RNA)得到富集。
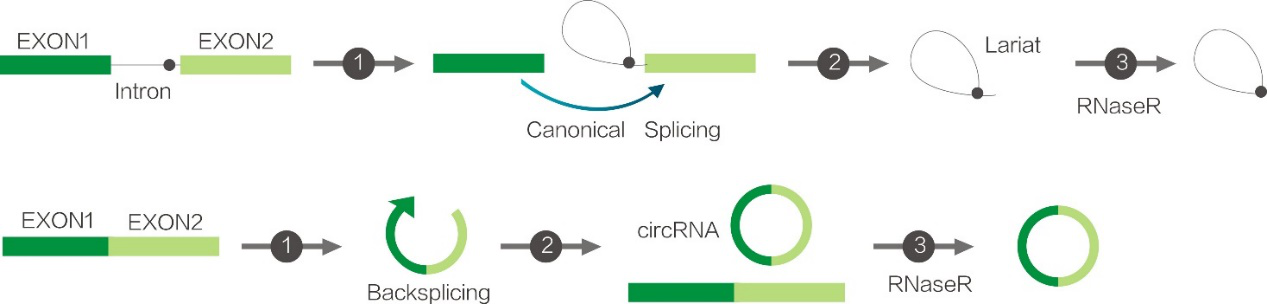
圖1.RNase R消化RNA示意圖
應用場景
1. circRNA的鑒定
根據RNase R(+)和RNase R(-)組中是否檢測到條帶來證明檢測的分子是否是circRNA。
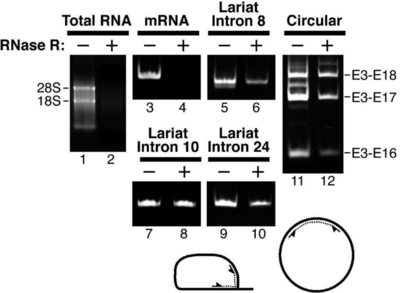
圖2.RNase R消化后RT-PCR檢測Lariat/Circular RNA
在圖2中,檢測mRNA在RNase R(-)中有條帶,在RNase R(+)中無條帶;檢測Lariat/Circular RNA,在RNase R(-)和RNase R(+)樣品中都有條帶,表明mRNA被消化,而Lariat/Circular RNA耐受消化(Suzuki H et al., 2006)。
2. circRNA的富集
高通量測序時常需要對circRNA進行富集,為探究RNase R消化后circRNA的富集程度和相關基因的變化,可以同時做RNase R(+)和RNase R(-)組樣品的測序。
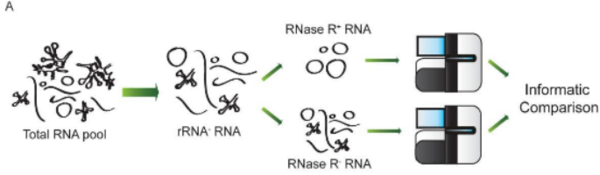
圖3.RNase R(+)和RNase R(-) RNA測序示意圖(Jeck WR et al., 2014)
有研究報道,測序顯示RNase R(+)組中Junction Reads相對于RNase R(-)樣本有5-10倍的富集,可鑒定出幾千到上萬個circRNAs(圖3)。
3.circRNA的純化
消除線性RNA殘余的干擾,是人工合成高純度circRNA的關鍵。高效的RNase R不僅是連接酶法circRNA人工合成的關鍵工具,在內含子自剪切的circRNA人工合成中也是必不可少的。
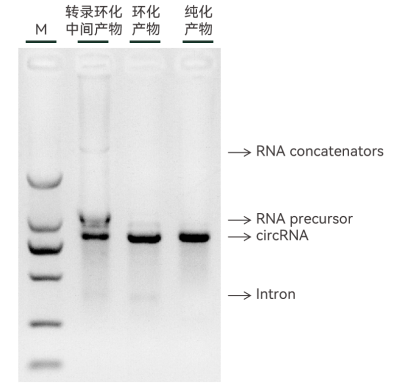
圖4. 吉賽生物circPrecise?平臺制備環狀RNA產物(“純化產物”使用GSPure? RNase R純化)
產品優勢
特異性強:特異性消化線性RNA;
高效快速:5-15min即可消化大部分線性RNA;
Buffer兼容:消化產物可直接用于下游實驗;
使用方便:體系簡單,37℃一步反應。
質量控制
SDS-PAGE 檢測純度>95%;Total RNA經RNase R消化后進行RT-qPCR檢測,線性 RNA豐度明顯降低,環形RNA豐度基本不變。
性能比較
1.RNA電泳檢測
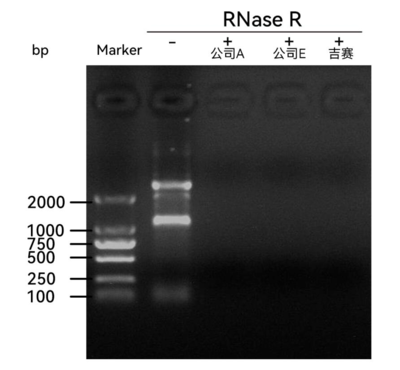
將10U RNase R加入到2.5 μg Total RNA中,于37℃孵育15 min后直接進行電泳檢測,結果顯示RNase R(+)組條帶變淡(不可見),表明RNase R對Total RNA產生消化作用。吉賽和公司A或公司E的RNase R對Total RNA消化效果相當。
3.RT-qPCR檢測
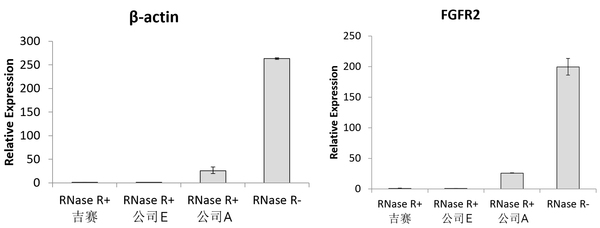
將10U RNase R加入到2.5 μg total RNA中,于37℃孵育30 min后進行RT-qPCR實驗,結果顯示RNase R+組β-actin和FGFR2的豐度都明顯降低,表明RNase R可消化線性RNA。吉賽和公司E的RNase R對線性RNA消化效果相當,優于公司A的RNase R。
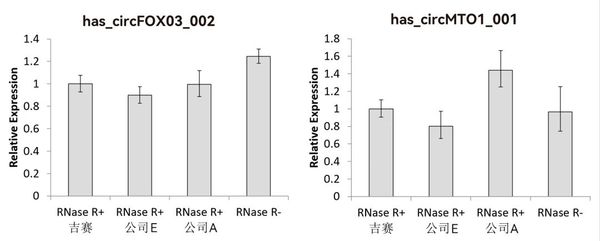
將10U RNase R加入到2.5 μg total RNA中,于37℃孵育30 min后進行RT-qPCR實驗,結果顯示RNase R+ 組中hsa_circFOXO3_002和hsa_circMTO1_001的豐度基本不變,表明circRNA耐受RNase R的消化。吉賽和公司E或公司A的RNase R效果相當。
注:數據計算以“RNase R+吉賽”組為對照,設定其相對豐度值為1。
案例1:

2022年7月21日,西北大學生命科學學院關鋒教授團隊在C1GALT1在膀胱癌中的生物功能及其調控機制的研究中取得了新進展,研究結果發表在Journal of Experimental & Clinical Cancer Research,題名“Dysregulation and prometastatic function of glycosyltransferase C1GALT1 modulated by cHP1BP3/ miR-1-3p axis in bladder cancer”。
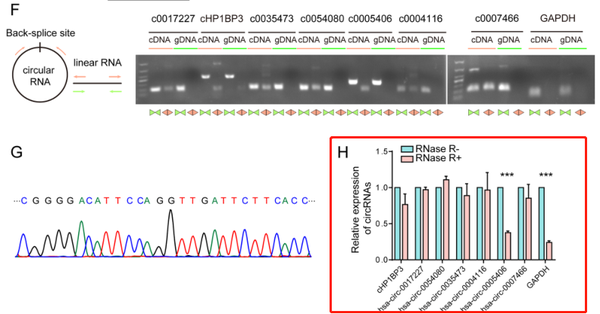
研究團結隊使用RNase R(R0301,吉賽生物)檢測了7個DEcircRs對RNase R消化的敏感性,以排除可能通過反式剪接、基因組重排或PCR產物產生的頭尾剪接。證明DEcircRs對RNase R的抗性均高于GAPDH對照。
案例2:

2022年8月12日,北京大學基礎醫學院張衛光教授團隊在Redox Biology雜志在線發表了題為“CircSV2b participates in oxidative stress regulation through miR-5107-5p-Foxk1-Akt1 axis in Parkinson's disease”的研究論文。該研究發現circRNA分子 circSV2b在帕金森病(Parkinson's disease,PD)發病過程中發揮著重要作用,并揭示了其作用機制。
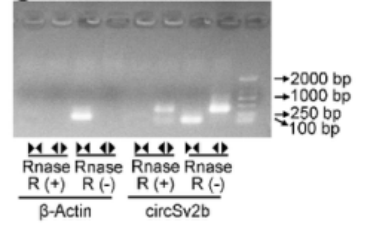
研究團結隊使用RNase R(R0301,吉賽生物)處理顯著降低線性SV2b和β-Actin的mRNA水平,而circSV2b的量未觀察到明顯的改變,證明circSV2b是一個高度穩定且內含子保留的環狀RNA。
[1] IF27.401 A novel protein AXIN1-295aa encoded by circAXIN1 activates the Wnt/β-catenin signaling pathway to promote gastric cancer progression. Molecular Cancer
[2] IF15.302 circPARD3 drives malignant progression and chemoresistance of laryngeal squamous cell carcinoma by inhibiting autophagy through the PRKCI-Akt-mTOR pathway. Molecular Cancer
[3] IF15.302 Circular RNA circCORO1C promotes laryngeal squamous cell carcinoma progression by modulating the let-7c-5p/PBX3 axis. Molecular Cancer
[4] IF13.493 The circFASN/miR-33a pathway participates in tacrolimus-induced dysregulation of hepatic triglyceride homeostasis. Signal Transduction and Targeted Therapy
[5] IF12.658 Dysregulation and prometastatic function of glycosyltransferase C1GALT1 modulated by cHP1BP3/ miR-1-3p axis in bladder cancer. JOURNAL OF EXPERIMENTAL & CLINICAL CANCER RESEARCH
[6] IF11.556 Engineering circular RNA regulators to specifically promote circular RNA production. Theranostics
[7] IF11.492 CircIMMP2L promotes esophageal squamous cell carcinoma malignant progression via CtBP1 nuclear retention dependent epigenetic modification. Clinical and Translational Medicine
[8] IF11.161 Circular RNA circ-MTHFD1L induces HR repair to promote gemcitabine resistance via the miR-615-3p/RPN6 axis in pancreatic ductal adenocarcinoma. JOURNAL OF EXPERIMENTAL & CLINICAL CANCER RESEARCH
[9] IF11.161 Warburg effect-promoted exosomal circ_0072083 releasing up-regulates NANGO expression through multiple pathways and enhances temozolomide resistance in glioma. JOURNAL OF EXPERIMENTAL & CLINICAL CANCER RESEARCH
[10] IF11.161 CircFAM73A promotes the cancer stem cell-like properties of gastric cancer through the miR-490-3p/HMGA2 positive feedback loop and HNRNPK-mediated β-catenin stabilization. JOURNAL OF EXPERIMENTAL & CLINICAL CANCER RESEARCH
[11] IF11.161 CircAGAP1 promotes tumor progression by sponging miR-15-5p in clear cell renal cell carcinoma. JOURNAL OF EXPERIMENTAL & CLINICAL CANCER RESEARCH
[12] IF10.392 A novel intronic circular RNA, circGNG7, inhibits head and neck squamous cell carcinoma progression by blocking the phosphorylation of heat shock protein 27 at Ser78 and Ser82. Cancer Communications
[13] IF10.122 Lipotoxicity-induced circGlis3 impairs beta cell function and is transmitted by exosomes to promote islet endothelial cell dysfunction. DIABETOLOGIA
[14] IF8.886 circRNA-0002109 promotes glioma malignant progression via modulating the miR-129-5P/EMP2 axis. Molecular Therapy-Nucleic Acids
[15] IF8.886 HnRNP-L-regulated circCSPP1/miR-520h/EGR1 axis modulates autophagy and promotes progression in prostate cancer. Molecular Therapy-Nucleic Acids
[16] IF8.886 Effects of long-term culture on the biological characteristics and RNA profiles of human bone-marrow-derived mesenchymal stem cells. Molecular Therapy-Nucleic Acids
[17] IF8.886 CircSTK40 contributes to recurrent implantation failure via modulating the HSP90/AKT/FOXO1 axis. Molecular Therapy-Nucleic Acids
[18] IF8.579 circPTCH1 promotes invasion and metastasis in renal cell carcinoma via regulating miR-485-5p/MMP14 axis. Theranostics
[19] IF8.469 Circular RNA hsa_circ_0043280 inhibits cervical cancer tumor growth and metastasis via miR-203a-3p/PAQR3 axis. Cell Death & Disease
[20] IF8.469 CircHAS2 promotes the proliferation, migration, and invasion of gastric cancer cells by regulating PPM1E mediated by hsa-miR-944. Cell Death & Disease
[21] IF8.469 Autophagy-related circRNA evaluation reveals hsa_circ_0001747 as a potential favorable prognostic factor for biochemical recurrence in patients with prostate cancer. Cell Death & Disease
[22] IF8.469 Circular RNA circACSL1 aggravated myocardial inflammation and myocardial injury by sponging miR-8055 and regulating MAPK14 expression. Cell Death & Disease
[23] IF8.469 Circular RNA circRUNX1 promotes papillary thyroid cancer progression and metastasis by sponging MiR-296-3p and regulating DDHD2 expression. Cell Death & Disease
1.RNase R消化成功的標準?
建議RNA經RNase R消化后取一部分直接進行電泳檢測,28、18S條帶變淡說明消化成功。(電泳結果比較直接,可觀測到消化成功,若不成功就是有問題)
2.RNase R實驗怎樣設計對照組?
參考下圖多幾個分組和對照,排除下影響因素。(增加多個對照,可以排除和找出異常情況的原因)
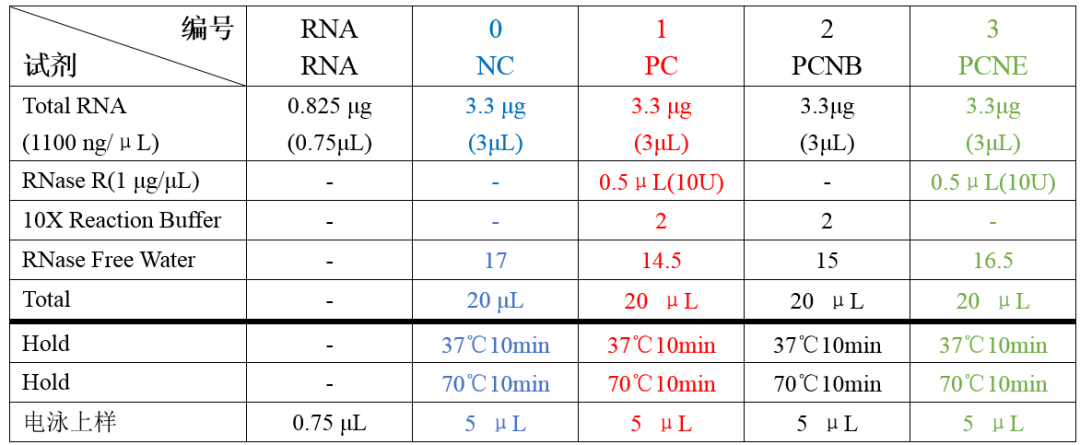
3.RNase R實驗的條件?
消化時間和RNase R用量可能需要預實驗摸索,找到合適的條件,讓線性RNA被消化而 circRNA保持不變。一般37℃消化15min,檢測內參的Ct值延后5~10都是比較成功的結果。(考慮消化不夠或過頭,或者結果數值不好看,可以做預實驗摸索)
4.RNase R引物檢測的問題?
注意檢測引物的特異性問題。注意區分circRNA和mRNA的同源序列。(引物有非特異擴增的時候,檢測結果數值完全不可能,任何說消化無效果或效果過頭都不可信)
5.CircRNA數量不變,算成功嗎?
總RNA被消化后,線性RNA數量減少,circRNA數量基本不變,則其中circRNA的比例增加了,即circRNA被富集了,后面qPCR檢測circRNA的豐度有可能會偏高,是正常的。
6. RNase R消化后如何回收RNA?
RNase R消化后消化液如果回收,圖方便可以使用Trizol回收,按說明書操作即可。如果不回收直接進行逆轉錄的,吸取消化液的體積不能超過逆轉錄體系的1/4,因為消化液中殘留的離子等會干擾下游的RT-qPCR反應。例如逆轉錄是20ul體系,則吸取的RNase R消化液不能超過5ul。
7.RNase R消化不成功怎么辦?
使用新的試劑盒或試劑重新提取RNA,保證RNA的純度和質量。(RNA的純度高才能消化效率高,酚類等殘留會明顯影響消化效果,gDNA污染也會減弱消化效果)
8. RNase R如何儲存
當天內使用的,RNase R(20U/ul)可以用無酶水稀釋,長期使用的,應該用Storage Buffer稀釋,配方在說明書里有。(用量少時體積小,有客戶想稀釋使用)
9.RNase R消化有什么注意事項
配置Nuclease-free的試劑,需要使用Nuclease-free的水、吸頭等試劑耗材,或者用DEPC處理過的水,另外實驗臺面(建議全程超凈臺內或生物安全柜內)也要用核酸酶清除劑噴霧和擦拭處理,這些都是做RNA實驗的配置。
10.RNase R消化的特殊情況
特殊情況:有的線性RNA耐受消化,有的circRNA也容易被消化。
11.RNase R消化后不用純化,直接跑膠,是否可以?
可以的。還能評估下消化效果,28、18S條帶變淡說明消化成功。
另,直接進行RT-PCR的,一般也可以不做純化,酶失活后直接進行逆轉錄反應即可。
12.消化反應中RNase R使用多少比較合適?
不同參考文章里RNase R的使用量和反應體系都有差別,最常見的是20 μL反應體系,使用比例為1-4 U RNase R/μg RNA,實驗中可能需要摸索調整。
13.RNase R消化反應溫度和時間設定多少?
RNase R消化一般于37℃進行反應,一般10-30 min即可消化掉大部分線性 RNA。常用5-15 min消化就能得到很好的結果。
14.RNase R消化反應后要不要滅活?
經RNase R消化后直接進行逆轉錄反應的,推薦在37℃反應結束后保持70℃ 10 min使RNase R滅活(也有65℃ 15min)。如果消化后要進行純化回收,也可以不做滅活。
15.RNase R消化鑒定
樣本RNase R消化前后取一部分直接進行電泳檢測,可直觀查看抽提的RNA質量及Rnase R消化效果,28、18S條帶變淡說明消化成功。若RNA純度不高,28S/18S條帶可能有凹型拖尾。若RNA有gDNA污染,可能殘留gDNA條帶。
16.RNase R消化反應條件?
不同的circRNA對RNase R的耐受性不同,具體反應時間需要自行摸索。一般反應時間為10-30 min,不推薦過長時間的消化。個別circRNA耐受RNase R消化力弱。對此,可以減少消化時間。
17.RNase R消化后需要測定產物濃度嗎?
不可直接測定RNA濃度,因為反應體系內的蛋白質、鹽離子等成分會導致RNA濃度測定不準確。建議先純化RNA再測定濃度。
18.RNase R消化后產物處理:
直接進行逆轉錄反應的,推薦RNase R滅活。滅活條件為:70℃保持15min。吸取消化液的體積不能超過逆轉錄體系的1/4,因為消化液中殘留的離子等會干擾下游的RT-qPCR反應。例如逆轉錄是20ul體系,則吸取的RNase R消化液不能超過5ul。
如果消化后要進行純化回收,也可以不做酶的失活。
填寫需求描述給我們
工具快速咨詢
400-8989-400
geneseed@geneseed.com.cn
- 返回頂部

 購物車
購物車




 廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓
廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn